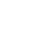质谱技术
MRM/PRM靶向定量蛋白质组学
靶向定量蛋白质组学技术,是充分利用质谱仪本身的特性,对进入质谱的离子进行多次筛选,进而靶向选定目标蛋白多肽的碎裂离子,进行定向检测的质谱技术。 目前,随着靶向定量蛋白质组学技术的发展,主要发展为MRM(多反应监测,Multiple Reaction Monitoring)和PRM(平行反应监测,Parallel Reaction Monitoring)两种技术路线。
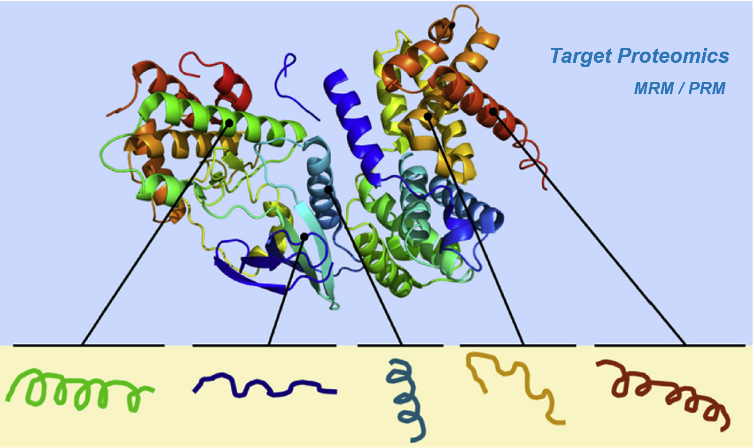
| MRM靶向蛋白质定量分析
MRM技术是利用三重四级杆质谱技术(QQQ)的定性多重离子筛选能力,开发出的对复杂样品中多个特定蛋白/肽段/ 小分子进行靶向定量分析的技术。Q1 筛选出与目标分子特异性一致的母离子;Q2 碰撞碎裂这些母离子,去除其它离子干扰;Q3 只对选定的MS2 离子对进行质谱信号采集,完成靶向定量。借助同位素标记的目标肽段,MRM 还可实现蛋白质的绝对定量。
| MRM靶向蛋白质定量分析
MRM技术是利用三重四级杆质谱技术(QQQ)的定性多重离子筛选能力,开发出的对复杂样品中多个特定蛋白/肽段/ 小分子进行靶向定量分析的技术。Q1 筛选出与目标分子特异性一致的母离子;Q2 碰撞碎裂这些母离子,去除其它离子干扰;Q3 只对选定的MS2 离子对进行质谱信号采集,完成靶向定量。借助同位素标记的目标肽段,MRM 还可实现蛋白质的绝对定量。
| PRM靶向蛋白质定量分析
PRM是依赖于高分辨/ 高精度质谱仪(尤其是obitrap系列)的高分辨率的优势而开发的靶向定量蛋白质分析技术,其优点是无需事先确定离子对和优化碰撞能量,操作简便,是MRM 的衍生技术。首先,利用四级杆质量分析器选择性地检测目标肽段的母离子;随后在Collision cell 中对母离子进行碎裂;最后利用Orbitrap 分析器在二级质谱中检测所选择的母离子窗口内的所有碎片信息,对目标蛋白/ 肽段进行定量分析。
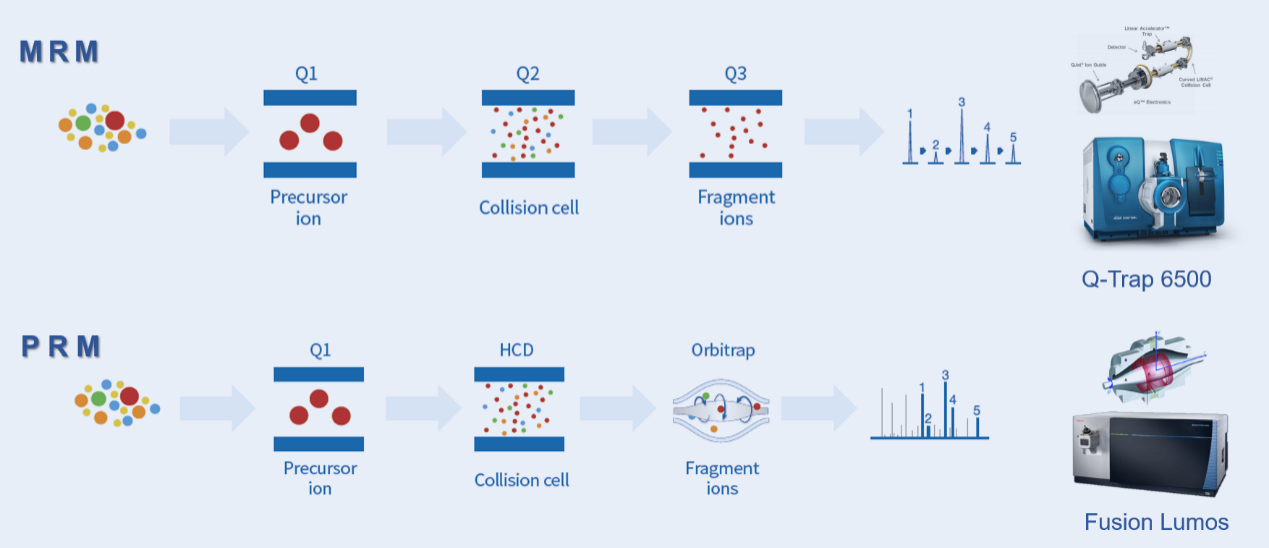
| 技术比较
无论MRM还是PRM均可较好的对非靶向蛋白质组筛选的数据进行靶向定量检测,由于两种技术开发的原理和依托的仪器平台不同。因此,两种技术的检测特点和具体应用均具有一定的差异。
MRM是在靶向分子检测的专用平台三重四级杆基础上开发的靶向大分子蛋白质检测技术,其特点是检测速度快,离子筛选能力强,并且配套的是微升液相系统,仪器稳定性好,定量准确性高。MRM技术尤其适合对特定小群目标蛋白进行大批量样本的定量检测(尤其是临床大队列样本)。
PRM是依托质谱仪的高分辨率性能开发的靶向大分子蛋白质检测技术,其特点是蛋白组的筛选和验证可以在一台质谱仪上实现,操作简便,检测成本低,可靶向监测的离子数量多。但是由于配套的是纳升液相系统,其仪器的稳定性较差一些。因此,一般是在非靶向蛋白质组学完成后短时期内,在质谱仪进行下一轮维护之前完成PRM实验。PRM技术适合小批量样本,较多目标蛋白靶向定量检测的实验需求。
| 经典案例

该文献比较了四种蛋白质组学方法(DDA、DIA、PRM、MRM)的性能。实验流程如下图,取来自三名欧洲人和两名非洲人的单个人类毛干(hair shafts)进行消化,从JPT Peptide Technologie公司获得24条遗传变异肽(GVP,genetically variant peptides)并将它们混合后添加到五名受试者的人类毛干蛋白溶液中,采用QE plus-DDA、Lumos-DIA、Lumos-PRM、QQQ-MRM进行分析。
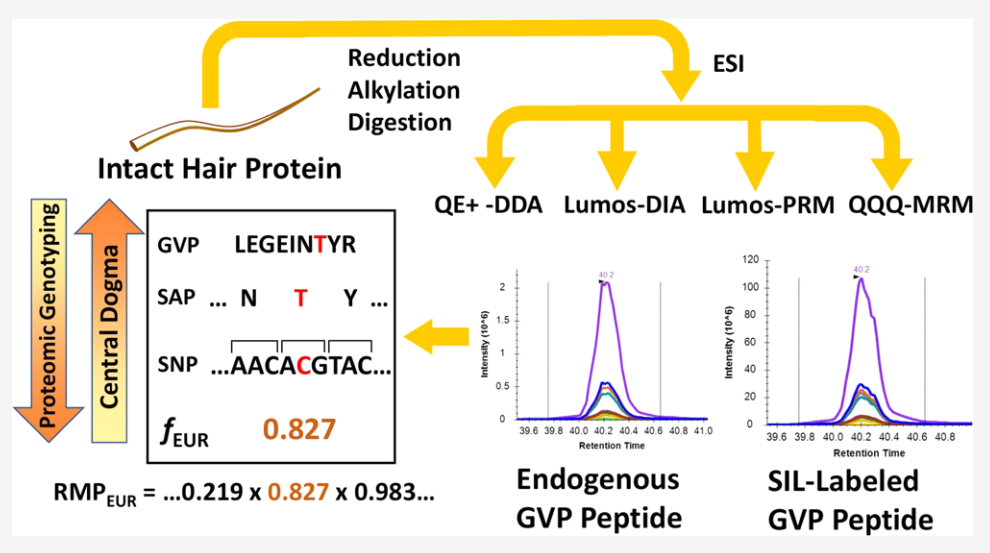
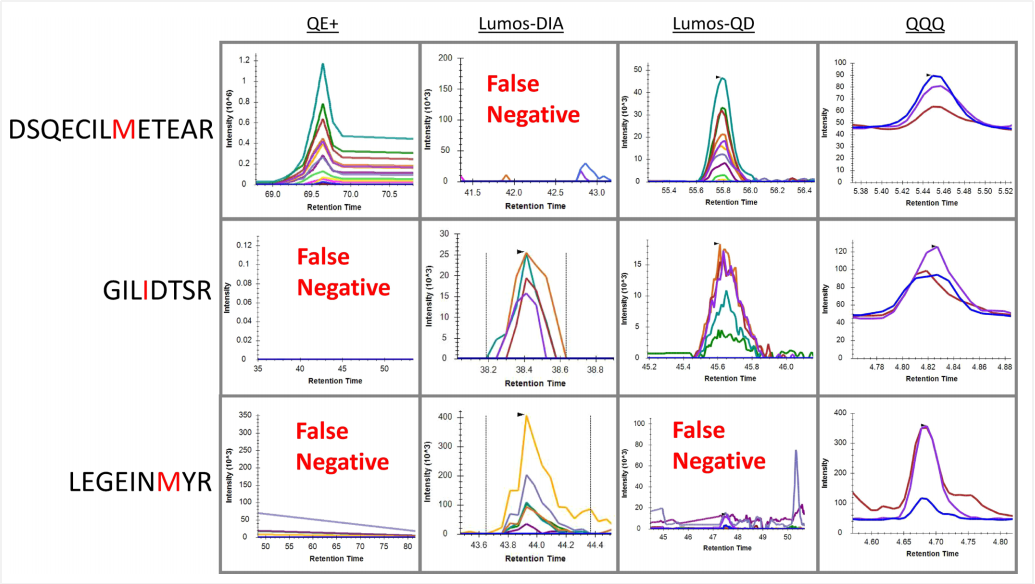
通过对四个分析平台的性能进行评估,选取24条肽段中的3条进行分析,发现第一条肽段DSQECILMETEAR在Lumos-DIA 中缺少,第二条肽段GILIDTSR在QE plus-DDA中缺少,第三条肽段LEGEINMYR在QE plus-DDA和Lumos-PRM中均缺少。总之这三条肽段MRM均能检出,而PRM只能检出其中的两条。
进一步对四个分析平台的性能进行综合性能评估,发现QE plus-DDA的鉴定百分比为26.4%,检测灵敏度为43.2%,错误发现率(FDR)最低,为2.6%。Lumos-DIA的鉴定百分比为13.2%,检测灵敏度为21.6%,FDR为5.0%,Lumos-PRM的鉴定百分比为33.3%,检测灵敏度为 54.5%,FDR也最高,为12.7%。QQQ-MRM的鉴定百分比为42.4%,检测灵敏度为69.3%,FDR为4.7%。
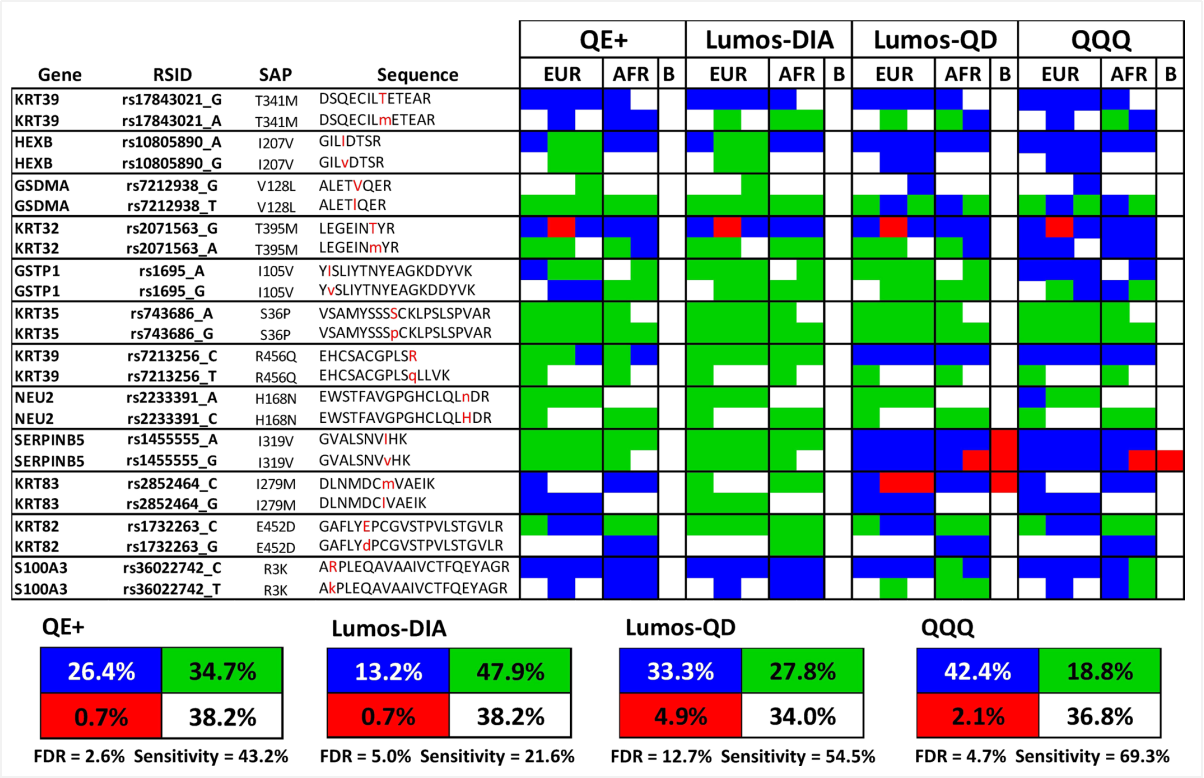
总体而言,与DIA相比,应用于PRM使检测灵敏度提高了1.3倍,MRM使检测灵敏度提高了1.6倍。与PRM相比,MRM的检测灵敏度较高,同时在FDR上,比PRM低。
| 相关文献
[1]. Zachary C Goecker, Kevin M Legg, Michelle R Salemi, et al. Alternative LC-MS/MS Platforms and Data Acquisition Strategies for Proteomic Genotyping of Human Hair Shafts. J Proteome Res. 2021 Oct 1;20(10):4655-4666.
[2]. Peggy Barschke, Patrick Oeckl, Petra Steinacker, et al. Different CSF protein profiles in amyotrophic lateral sclerosis and frontotemporal dementia with C9orf72 hexanucleotide repeat expansion. J Neurol Neurosurg Psychiatry. 2020 May;91(5):503-511.
[3]. Megha Bhardwaj, Korbinian Weigl, Kaja Tikk, et al. Multiplex quantitation of 270 plasma protein markers to identify a signature for early detection of colorectal cancer. Eur J Cancer. 2020 Mar;127:30-40.
[4]. Patrick Oeckl, Patrick Weydt, Dietmar R Thal, et al. Proteomics in cerebrospinal fluid and spinal cord suggests UCHL1, MAP2 and GPNMB as biomarkers and underpins importance of transcriptional pathways in amyotrophic lateral sclerosis. Acta Neuropathol. 2020 Jan;139(1):119-134.
[5]. Gerard A Silvestri, Nichole T Tanner, Paul Kearney, et al. Assessment of Plasma Proteomics Biomarker's Ability to Distinguish Benign From Malignant Lung Nodules: Results of the PANOPTIC (Pulmonary Nodule Plasma Proteomic Classifier) Trial. Chest. 2018 Sep;154(3):491-500.
[6]. Anil Vachani, Harvey I Pass, William N Rom, et al. Validation of a Multiprotein Plasma Classifier to Identify Benign Lung Nodules. J Thorac Oncol. 2015 Apr;10(4):629-37.

地址:上海市闵行区绿洲环路396弄5幢4层
电话:400-869-2936,021-33968791
邮箱:support@wayenbiotech.com
传真:021-33938792
QQ:2120485725
战略合作
战略合作
| Full Moon | RayBiotech | CDI | IZON | Bio-Rad | R&D Systems | IsoPlexis | Akoya | 南模生物 | 欧易生物 | 上海实验动物研究中心 | 美吉生物 | 麦特绘谱 |
特别说明:本网所有网页均为华盈生物原创信息,转载必须征求华盈生物同意,并同时注明本网名称及网址。
专利代码